基因组数据整合提高了苹果果实性状的预测准确性
在过去的几十年里,世界见证了基因组分析工具的巨大进步。虽然这些工具通常与生物学和医学领域联系在一起,但它们在农业领域也非常有价值。利用从下一代测序技术获得的大量 DNA 标记,育种者可以进行基因组预测,并根据预测的性状值选择有前途的个体。
旨在提高水果品质的各种系统和方法都使用基因分析。其中之一包括基因选择 (GS) 和基因预测 (GP)。这种现代育种方法使用统计模型根据先前收集的基因组及其相关特征来评估特定个体的整个基因图谱。这使育种者能够预测未来幼苗阶段将产生的水果特征。相比之下,全基因组关联研究 (GWAS) 则专注于寻找导致特定水果特征的确切基因变异。
到目前为止,GP 和 GWAS 主要使用来自单一系统的 DNA 标记,当使用的系统过时时,必须使用更新的系统重新分析。然而,很难重新分析在以前的系统中分析过的果树育种选择种群,因为不可能从选择过程中丢弃的个体中重新获得 DNA。因此,在2024 年 7 月8 日发表在《园艺研究》上的一项最新研究中,由日本千叶大学高级学术研究所副教授 Mai F. Minamikawa 领导的研究小组着手阐明在进行 GP 和 GWAS 时,结合来自不同系统的苹果数据是否可以产生更准确的结果。该团队的其他成员包括日本国家农业和食品研究组织果树和茶科学研究所的 Miyuki Kunihisa 博士和日本东京大学农业与生命科学研究生院的 Hiroyoshi Iwata 教授。
首先,研究人员将从两种不同的基因分型系统(即 Infinium 和直接随机扩增子测序 (GRAS-Di))获得的苹果数据集组合起来。然后,他们使用这些组合的基因型标记对总共 24 种不同的水果性状进行 GP 和 GWAS,包括酸度、甜度、收获时间和固体可溶物含量。该团队比较了使用单独或两者结合训练的数据集进行预测的性能。
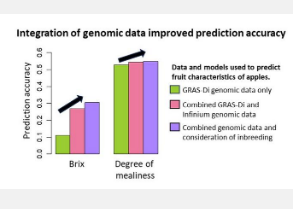
结果非常令人鼓舞;当使用 Infinium 和 GRAS-Di 组合数据集对多种水果性状进行分析时,基因组预测的准确性和 GWAS 系统的检测能力显著提高。这表明,将来自不同系统的数据结合起来并利用历史数据是有好处的。
为了进一步突破极限,研究人员还训练了 GP 模型,使其考虑了近亲繁殖的影响。有趣的是,这些结果还暗示了组合方法对某些性状表现更好,包括白利度和粉度。不过,这些发现还不太具有决定性,正如 Minamikawa 博士所说,“尽管近亲繁殖数据可以提高苹果果实性状 GS 的准确性,但仍需要进一步研究以了解果实性状与近亲繁殖之间的关系。 ”
总体而言,这项研究的结果暗示了一种通过利用现有数据集来提高 GS 和 GWAS 准确性的便捷方法。这可能对农业产生许多积极影响,正如 Minamikawa 博士所强调的那样,“通过在幼苗阶段使用高精度 GS 从众多个体中识别出优良基因型并使用精确的 GWAS 检测目标性状的遗传变异,可以解决诸如植物体型大和果树幼苗期长等挑战。 ”
免责声明:本答案或内容为用户上传,不代表本网观点。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。 如遇侵权请及时联系本站删除。
